비지도학습 :
데이터가 주어질 때, 데이터에 대한 정답이 주어지지 않음
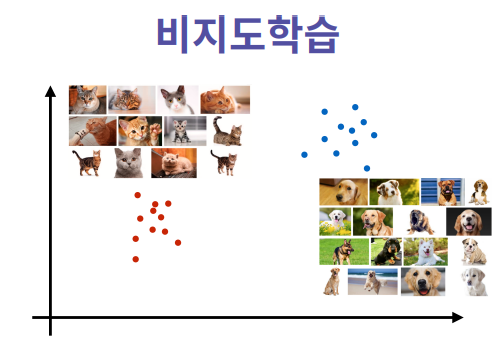
차원축소를 통해 모델에서 Clustring을 활용하며 어떤 데이터의 포인트에 답이나 레이블 없이 데이터 의 군집을 찾아낸다

60% 강아지 40% 고양이의 경우는 없음, 확률적으로 이를 보일 수 있지만 현실세계에서는 그렇진 않다.
Hard clustering은 선을 그어서 이를 분류한다.

각각의 클래스가 조금씩 0이나 1로 섞여있다, 라고 할때 이를 soft clustring이라고 한다.

자연적으로 데이터가 일어난다고 하고 클래스가 지나갈 때, 점차적으로 움직이게 된다.
Softclustring을 사용하지 못할때, HardClustring을 사용함

HardClustering 은 비슷한 데이터 포인트 끼리 모으는데, 비슷하게 뭉친 클러스터 2개를 찾는다고 할 때, 컴퓨터는 서로 비슷한 것으로 찾을 수 있다
각각의 데이터 포인트 안에 비슷한 것끼리 뭉치는 것이다.
사람이 보고 이를 판단할 수 있어야 한다.
컴퓨터는 A, B 의 클래스를 구분할 수 있어야 한다.

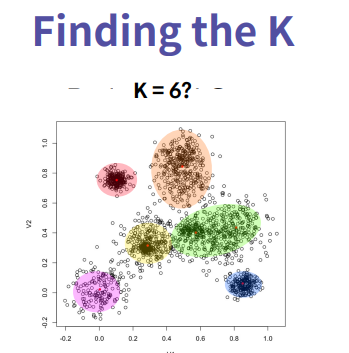
K 즉 몇개의 군집을 찾을건지 찾는것도 매우 중요하다. K를 데이터를 잘 설명하는 K 갯수가 몇개 일지에 따라 클러스터가 다르게 구분된다. 설정을 고려할 때, 데이터를 만드는 소스가 어떤 특성을 갖고있는지 사람의 몸무게 라고 햇을 때, 데이터가 남성과 여성 두개로 나뉠경우 남성의 몸무게가 여성보다 높으므로 데이터의 특성을 알경우, 분석 결과를 알 수 있다.
K=2를 몇개로 정할 경우 일지에 따라 사전지식이 있다면 K를 정하는데 수월할 것이다.


PCA
도메인에 대해 잘 모를 경우, 차원축소를 사용하여 구분을 할 수 있다. PCA는 대표적인 차원축소 알고리즘이며, 주성분 분석이라고 불린다.
사용하는 이유는 시각화를 위해서 각각의 차원을 눈으로 보기 위해 2차원으로 줄일때,PCA를 사용한다.
데이터의 손실을 최소화 하는게 PCA의 역할이다.

데이터가 3차원으로 퍼져있다고 볼 때, 시각화의 정제를 사용할 수 있다.
타 클러스터링과 다른 방식은 차원은 크게 중요하지 않고 정제하는데 많이
사용하게 된다.
주성분 분석(Principal Component Analysis, PCA)
주성분 분석(Principal Component Analysis, PCA)은 고차원 데이터를 저차원의 데이터로 변환하는 알고리즘
13차원의 와인 데이터셋을 2차원으로 변환
import sklearn.decomposition
import matplotlib.pyplot as plt
import numpy as np
import csv
def main():
X, attributes = input_data()
pca_array = normalize(X)
pca, pca_array = run_PCA(X, 2)
visualize_2d_wine(pca_array)
print(X)
def input_data():
X = []
attributes = []
with open("data/attributes.txt") as fp:
attributes = fp.readlines()
attributes = [x.strip() for x in attributes]
print(attributes)
csvreadr = csv.reader(open("data/wine.csv"))
for line in csvreadr:
float_line = [float(x) for x in line]
X.append(float_line)
return np.array(X), attributes
def run_PCA(X, num_components):
pca = sklearn.decomposition.PCA(n_components = num_components)
pca.fit(X)
pca_array = pca.transform(X)
return pca, pca_array
def normalize(X):
for i in range(X.shape[1]):
X[:,i] = X[:,i] - np.min(X[:,i])
X[:,i] = X[:,i] / np.max(X[:,i])
'''
각각의 feature에 대해,
178개의 데이터에 나타나는 해당하는 feature의 값이 최소 0, 최대 1이 되도록
선형적으로 데이터를 이동
'''
return X
def visualize_2d_wine(X):
plt.scatter(X[:,0],X[:,1])
'''X를 시각화하는 코드를 구현'''
plt.savefig("image.png")
elice_utils.send_image("image.png")
if __name__ == '__main__':
main()
K-means 클러스터링
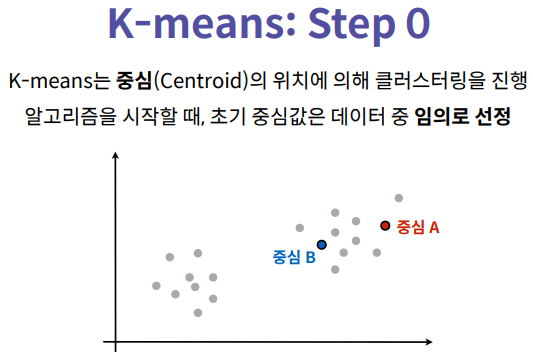
K-means는 반복을 이용한 알고리즘이며, 한번의 스텝을 이해하면
중심을 기반으로 각 클러스터의 중심과의 거리를 의미하며, 중심이라는 것은 데이터 포인트가 있다고 할 때, 클러스터 A와 B가 있으면 A에서의 중심은 A안에 있는 데이터 포인트의 X좌표의 평균값을 구하고
Y좌표의 평균값을 구하여 중심과의 거리를 구해서 데이터 포인트가 A포인트와 거리, B포인트와 거리를
X에서 C를 뺀것의 Norm을 구한다.


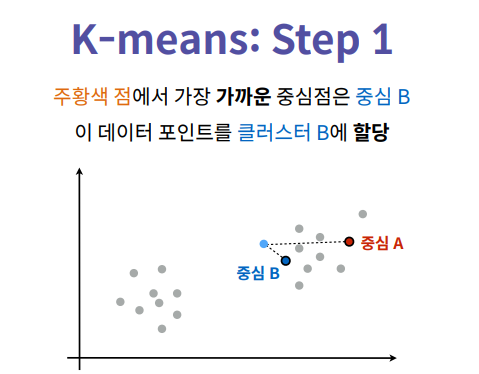
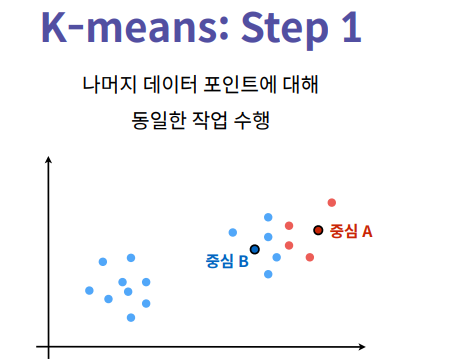




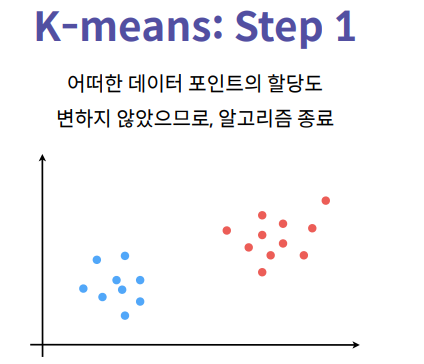
K-Means 클러스터링
2차원으로 줄여 시각화한 와인 데이터를 이용해 어떤 와인이 비슷한지 알아내고, 비슷한 와인을 묶는 알고리즘을 작성
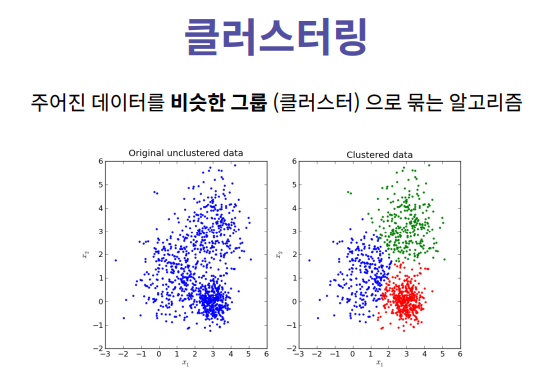
클러스터링, 또는 클러스터 분석은 주어진 개체에서 비슷한 개체를 선별하고 묶는(grouping) 작업을 의미
또한, 대푯값을 추출하여 각 클러스터를 대표하는 개체를 찾아낼 수 있음
K-Means 클러스터링은 주어진 데이터를 K개의 클러스터로 묶는 알고리즘
알고리즘은 어떠한 선행 학습 없이 자동으로 클러스터를 분류하고 개체들의 묶음을 추출
K의 개수를 조정하면 클러스터의 일반도를 조정할 수 있습니다. 생물 데이터에서, K=2 인 경우에는 동물과 식물 클러스터가 나올 가능성이 높음
K가 매우 크다면, 동물/식물 내의 세부 분류, 강/목/속/종 등의 분류가 나올 수 있습니다. K-means는 완전한 자율 알고리즘이기 때문에 K를 제외한 다른 입력값이 없으며, random 값을 사용하므로 여러 번을 반복 실행하여 평균 성능을 측정하는 것이 일반적
import sklearn.decomposition
import sklearn.cluster
import matplotlib.pyplot as plt
import numpy as np
def main():
X, attributes = input_data()
X = normalize(X)
pca, pca_array = run_PCA(X, 2)
labels = kmeans(pca_array, 5, [0, 1, 2,3,4])
visualize_2d_wine(pca_array, labels)
def input_data():
X = []
attributes = []
with open('data/wine.csv') as fp:
for line in fp:
X.append([float(x) for x in line.strip().split(',')])
with open('data/attributes.txt') as fp:
attributes = [x.strip() for x in fp.readlines()]
return np.array(X), attributes
def run_PCA(X, num_components):
pca = sklearn.decomposition.PCA(n_components=num_components)
pca.fit(X)
pca_array = pca.transform(X)
return pca, pca_array
def kmeans(X, num_clusters, initial_centroid_indices):
import time
print(X)
N = len(X)
centroids = X[initial_centroid_indices]
labels = np.zeros(N)
while True:
'''
Step 1. 각 데이터 포인트 i 에 대해 가장 가까운
중심점을 찾고, 그 중심점에 해당하는 클러스터를 할당하여
labels[i]에 넣습니다.
가까운 중심점을 찾을 때는, 유클리드 거리를 사용합니다.
미리 정의된 distance 함수를 사용합니다.
'''
is_changed = False
for i in range(N):
distances = []
for k in range(num_clusters):
# X[i] 와 con
k_dist = distance(X[i], centroids[k])
distances.append(k_dist)
if labels[i] != np.argmin(distances):
is_changed = True
labels[i] = np.argmin(distances)
'''
Step 2. 할당된 클러스터를 기반으로 새로운 중심점을 계산합니다.
중심점은 클러스터 내 데이터 포인트들의 위치의 *산술 평균*
으로 합니다.
'''
for k in range(num_clusters):
x = X[labels == k][:,0]
y = X[labels == k][:,1]
x = np.mean(x)
y = np.mean(y)
centroids[k] = [x,y]
print(centroids)
'''
Step 3. 만약 클러스터의 할당이 바뀌지 않았다면 알고리즘을 끝냅니다.
아니라면 다시 반복합니다.
'''
if not is_changed:
break
return labels
def distance(x1, x2):
return np.sqrt(np.sum((x1 - x2) ** 2))
def normalize(X):
for dim in range(len(X[0])):
X[:, dim] -= np.min(X[:, dim])
X[:, dim] /= np.max(X[:, dim])
return X
'''
이전에 더해, 각각의 데이터 포인트에 색을 입히는 과정도 진행합니다.
'''
def visualize_2d_wine(X, labels):
plt.figure(figsize=(10, 6))
plt.scatter(X[:,0], X[:,1], c=labels)
plt.savefig("image.svg", format="svg")
if __name__ == '__main__':
main()'파이썬 이것저것 > 파이썬 머신러닝' 카테고리의 다른 글
| [Python] 다중, 다항회귀분석 (0) | 2022.07.18 |
|---|---|
| [Python] 머신러닝 단순선형회귀분석 (0) | 2022.07.18 |
| [파이썬] XGB 활용하여 성적예측 (0) | 2022.04.23 |